MyoMapNet
Guo R, El-Rewaidy H, Assana S, et al. Accelerated cardiac T1 mapping in four heartbeats with inline MyoMapNet: a deep learning-based T1 estimation approach. Journal of Cardiovascular Magnetic Resonance, 2022, 24(1): 6.
提出目的
为了缩短T1map整体采样时间,减少了序列中采集的图像,并用全连接网络进行参数估计。
原始的MOLLI3(3)3(3)5采集模板需要17个心跳周期的屏气时间进行采集。MOLLI5(3)3和MOLLI4(1)3(1)2需要11个心跳周期的屏气时间。MyoMapNet通过类似MOLLI的序列,也通过翻转恢复脉冲进行信号翻转,通过bSSFP进行采集,但只采集4张图,所以仅需要4个心动周期的屏气时间,基本上可以在一次屏气就完成采集。
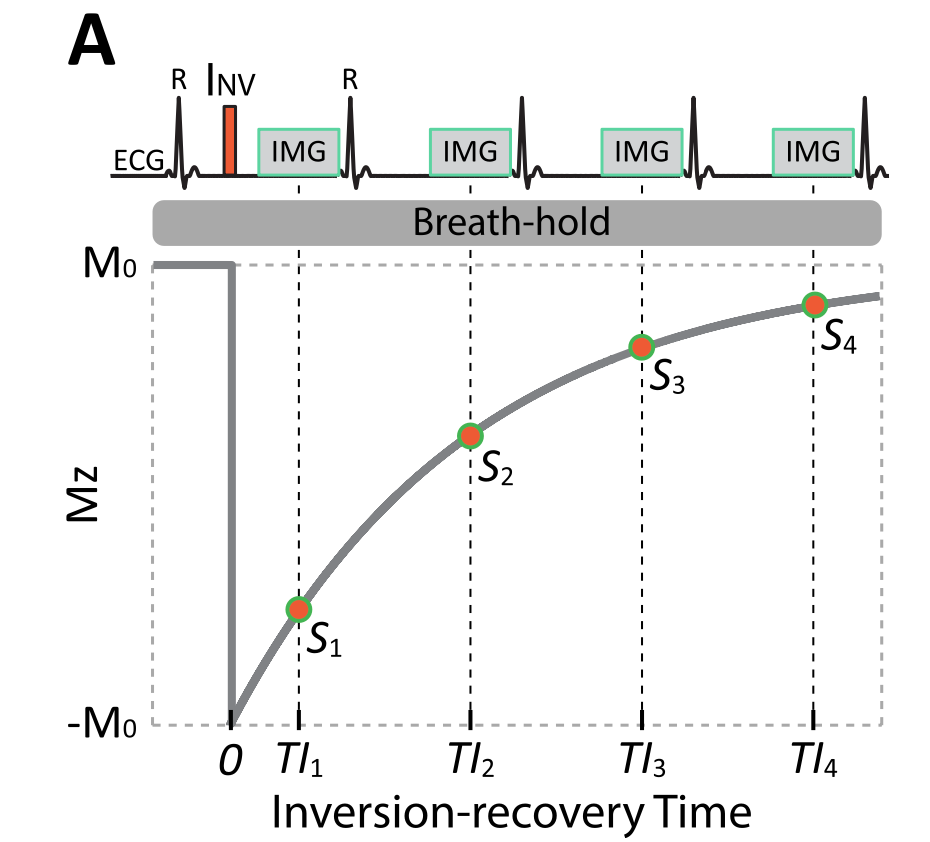
神经网络结构
神经网络采用全连接结构,有开源代码。
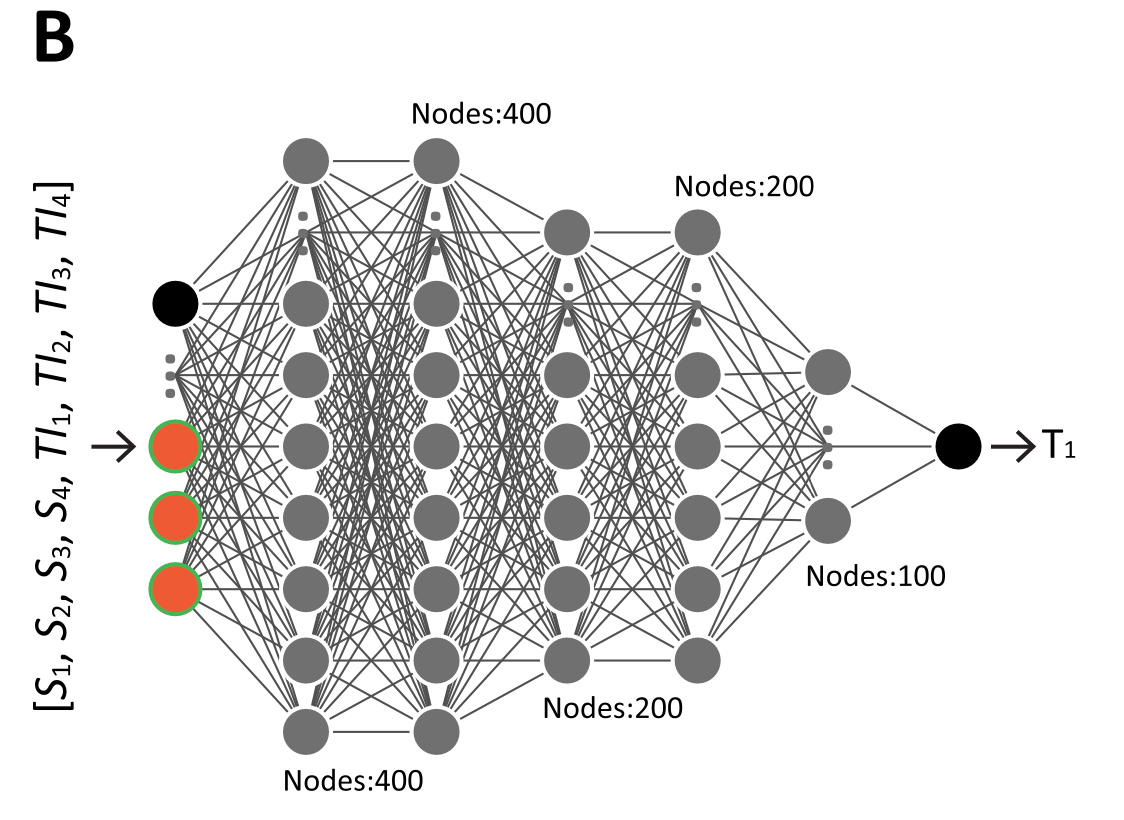
网络没有什么特殊的结构,网络的输入是采集到的信号$S_i$和对应的翻转时间$TI_i$,输出是最终的T1。没有考虑三参数模型和Look-Locker参数修正,而是直接输出最终的T1。
数据集和损失函数
数据集使用真实采集的749例病人数据,非公开数据集。采集机器是西门子的Vida。数据中包含常规T1和造影剂增强T1。运动修正采用机器自带的算法。T1是通过3参数模型加LL修正的方法自己拟合的。文中没有说用了什么拟合方法,应该是最小二乘法。
在训练过程中,采用传统方法拟合的T1值作为label,采集到的信号和翻转时间作为输入。损失函数采用MAE。 $$ \text { MAE } =\frac{1}{\text { Number of Pixels }} \mid\text { MOLLI T }_1-\text { Myo MapNet } \mathrm{T}_1 \mid $$
结果验证
通过对体模和人体数据的拟合结果进行统计分析,来验证所提的方法是否和传统MOLLI计算的T1值一致。
统计分析采用Bland–Altman分析法。